Roche uPath PD-L1 image analysis for NSCLC IVD Algorithm Benutzerhandbuch
- Typ
- Benutzerhandbuch

Algorithmus-Handbuch für die uPath
PD-L1 (SP263) Bildanalyse bei
nicht-kleinzelligem Lungenkarzinom


Inhaltsverzeichnis
Einleitung 1
Zusammenfassung und Erklärung des Algorithmus 2
Verwendungszweck 3
Verwendungszweck des Produkts 3
Zweck dieses Algorithmus-Handbuchs 3
Klinische Bedeutung 4
Testprinzip 5
Einschränkungen 6
Datensicherheit 7
Arbeitsablauf mit dem uPath PD-L1 Bildanalyse-Algorithmus für NSCLC 8
Arbeitsablauf für Pathologen 9
PD-L1-Bildanalyse 12
Bewertung der PD-L1-Färbung 15
Bewertung der Färbung mit dem PD-L1-Algorithmus 15
Färbungsmerkmale 15
Leistungsmerkmale 26
Methodenvergleich 26
Pathologen-Reproduzierbarkeitsstudien 27
Scanner-Reproduzierbarkeitsstudien 28
Fehlerbehebung 29
Literatur 33


Einleitung
Die Roche uPath enterprise software (uPath enterprise software)
mit dem uPath PD-L1 (SP263) Bildanalyse-Algorithmus für
nicht-kleinzelliges Lungenkarzinom (Non-Small Cell Lung
Cancer, NSCLC) (uPath PD-L1 (SP263) Bildanalyse-Algorithmus
für NSCLC) ist ein Softwaresystem zur Unterstützung
der quantitativen Auswertung der Proteinexpression in
immunhistochemisch gefärbten (IHC) Schnitten von
formalinfixiertem, paraffineingebettetem (FFPE) normalem und
neoplastischem Gewebe.
Die uPath enterprise software ist eine digitale Software-
Komplettlösung für die Pathologie zur Erfassung, Verwaltung,
Ansicht, Analyse, Weitergabe und Befunderstellung digitaler
Darstellungen von Pathologieproben. Mit der uPath enterprise
software können digitale Bilder in verschiedenen Vergrößerungen
dargestellt, Anmerkungen zugewiesen, Gewebeschnitte
vermessen, Bildanalysen durchgeführt und Berichte erstellt
werden.
Hinweis: Der uPath PD-L1 (SP263) Bildanalyse-Algorithmus für
NSCLC ist eine ergänzende, computergestützte Methode zur
Erfassung und Vermessung von mikroskopischen Bildern von
IHC-gefärbten Gewebeproben auf Objektträgern zum Nachweis
von PD-L1-Protein. Es ist Aufgabe des Pathologen, die Validität
der Bildanalyse-Scores durch die Analyse passender Kontrollen
zu verifizieren, wie im Methodenblatt für den VENTANA PD-L1
(SP263) Rabbit Monoclonal Primary Antibody (erhältlich unter
www.ventana.com) beschrieben wird.
Algorithmus-Handbuch für die uPath PD-L1 (SP263) Bildanalyse bei nicht-kleinzelligem Lungenkarzinom (Non-Small Cell Lung Cancer, NSCLC) 1

2 Algorithmus-Handbuch für die uPath PD-L1 (SP263) Bildanalyse bei nicht-kleinzelligem Lungenkarzinom (Non-Small Cell Lung Cancer, NSCLC)
Zusammenfassung und Erklärung des
Algorithmus
Für die Bildanalyse mit der uPath enterprise software werden
eine oder mehrere Zielregionen (ROIs) ausgewählt und markiert.
Jede ROI kann in verschiedenen Vergrößerungen angezeigt und
mithilfe des uPath PD-L1 (SP263) Bildanalyse-Algorithmus für
NSCLC analysiert werden. Die Gesamtzahl der Ziel-Tumorzellen
(TC) wird quantifiziert, und die TC werden ihrem Färbestatus
(gefärbt oder nicht gefärbt) entsprechend stratifiziert. Der uPath
PD-L1 (SP263) Bildanalyse-Algorithmus für NSCLC dividiert die
Zahl der gefärbten TC durch die Gesamtzahl der TC (gefärbt
und nicht gefärbt), um einen Score für PD-L1-TC-Positivität auf
einer Skala von 0–100% zu ermitteln. Der uPath PD-L1 (SP263)
Bildanalyse-Algorithmus für NSCLC kann einen Score für eine
bestimmte ROI oder einen zusammenfassenden Score für alle
gewählten ROIs auf dem jeweiligen Objektträger erstellen.
Obwohl bei der Gesamtanalyse mit dem Algorithmus auch
Nicht-Tumorzellen erfasst werden, werden im Overlay und als
Ergebnis nur die TC gezeigt, die bei der Berechnung des TC-
Positivitäts-Scores verwendet werden. Der Pathologe kann den
vom uPath PD-L1 (SP263) Bildanalyse-Algorithmus für NSCLC
vorgeschlagenen Score übernehmen oder den vorgeschlagenen
Score mit einem anderen Score überschreiben. Der Pathologe
muss sorgfältig überprüfen, welche TC von dem Algorithmus
als positiv und negativ markiert worden sind, und das Ergebnis
als korrekt bestätigen oder eine manuelle Überschreibung
vornehmen. Der uPath PD-L1 (SP263) Bildanalyse-Algorithmus
für NSCLC führt keine unabhängige Dateninterpretation
durch und sollte daher nur von qualifizierten Pathologen und
im Zusammenhang mit den histologischen Untersuchungen,
relevanten klinischen Daten und geeigneten Kontrollen
angewendet werden. Er ist zur Unterstützung des Pathologen bei
der Bewertung der PD-L1-Expression bei 50% bestimmt.

Verwendungszweck
Zweck dieses Algorithmus-Handbuchs
Dieses Algorithmus-Handbuch für die uPath PD-L1 (SP263)
Bildanalyse bei NSCLC (Algorithmus-Handbuch) dient folgenden
Zwecken:
• Bereitstellung von Basisinformationen zum
Verwendungszweck des uPath PD-L1 (SP263)
Bildanalyse-Algorithmus für NSCLC, zu Testprinzipien und
Testbeschränkungen.
• Beschreibung der notwendigen Anforderungen an
Materialien, IT, Datensicherheit und Netzwerk.
• Ausführliche Schritt-für-Schritt-Beschreibung der
Verwendung des uPath PD-L1 (SP263) Bildanalyse-
Algorithmus für NSCLC.
• Darstellung der korrekten Verwendung des uPath PD-L1
(SP263) Bildanalyse-Algorithmus für NSCLC anhand von
Bildmaterial.
• Bereitstellung eines Tools für Pathologen zur Verwendung des
uPath PD-L1 (SP263) Bildanalyse-Algorithmus für NSCLC mit
Schnitten von FFPE-NSCLC-Gewebe nach Anfärbung mit dem
VENTANA PD-L1 (SP263) Assay.
• Exemplarische Darstellung von Bildmaterial zur Verwendung
des uPath PD-L1 (SP263) Bildanalyse-Algorithmus für NSCLC
bei schwierigen Fällen.
• Beschreibung der Leistungsmerkmale des uPath PD-L1
(SP263) Bildanalyse-Algorithmus für NSCLC.
Verwendungszweck des Produkts
Der uPath PD-L1 (SP263) Bildanalyse-Algorithmus für
NSCLC unterstützt Pathologen beim Nachweis und der
Semiquantifizierung von PD-L1-Protein in formalinfixiertem,
paraffineingebettetem NSCLC-Gewebe. Bei Verwendung mit
dem VENTANA PD-L1 (SP263) Assay ist er als Unterstützung bei
der Identifizierung von Patienten mit NSCLC zur Behandlung mit
Therapien bestimmt, bei denen der zugelassenen Fachinformation
des therapeutischen Produkts entsprechend für TC ein PD-L1-
Positivitätsgrenzwert von ≥50% besteht.
Hinweis: Die uPath PD-L1 (SP263) Bildanalyse für NSCLC ist eine
ergänzende, computergestützte Methode zur Erfassung und
Vermessung von mikroskopischen Bildern von IHC-gefärbten
Gewebeproben auf Objektträgern zum Nachweis von PD-
L1-Protein. Es ist Aufgabe des Pathologen, die Validität der
Bildanalyse-Scores durch die Analyse passender Kontrollen
zu verifizieren, wie im Methodenblatt für den VENTANA PD-L1
(SP263) Assay (Bestell-Nr. 741-4905) beschrieben wird.
Dieser Algorithmus ist für die Verwendung in der In-vitro-
Diagnostik (IVD) bestimmt.
Algorithmus-Handbuch für die uPath PD-L1 (SP263) Bildanalyse bei nicht-kleinzelligem Lungenkarzinom (Non-Small Cell Lung Cancer, NSCLC) 3

Klinische Bedeutung
Seit mehreren Jahrzehnten ist Lungenkrebs weltweit die am
häufigsten auftretende Krebsart und die häufigste mit Krebs
zusammenhängende Todesursache. Schätzungen zufolge handelt
es sich bei 12,9% aller neuen Krebsfälle um Lungenkrebs, und pro
Jahr sind fast 1,76Millionen Todesfälle weltweit auf diese Krankheit
zurückzuführen. Dies entspricht ungefähr einem Fünftel der Todesfälle
im Zusammenhang mit Krebs. Das nicht-kleinzellige Lungenkarzinom
(Non-Small Cell Lung Cancer, NSCLC) ist die häufigste Form von
Lungenkrebs und macht ungefähr 85% aller Fälle aus. Bei den
meisten Patienten mit NSCLC ist die Erkrankung zum Zeitpunkt
der Vorstellung beim Arzt inoperabel und lokal fortgeschritten
(StadiumIIIB) oder metastasiert (StadiumIV). In beiden Fällen gibt
es derzeit keine kurativen Behandlungsmöglichkeiten. Die relative
Überlebensrate nach 5 Jahren bei diagnostiziertem NSCLC im
fortgeschrittenen Stadium liegt bei 4,7%.
PD-L1 (Programmed Death-Ligand 1) ist ein auf aktivierten
T-Zellen exprimiertes Transmembranprotein, das die Immunantwort
herunterreguliert, indem es an seine zwei inhibitorischen
Rezeptoren PD-1 (Programmed Death-1) und B7-1 (CD80) bindet.
Die Bindung von PD-L1 an PD-1 hemmt die T-Zellproliferation,
Zytokinproduktion und zytolytische Aktivität, was zur funktionellen
Inaktivierung oder Erschöpfung von T-Zellen führt. PD-L1 bindet
außerdem an CD80 auf antigenpräsentierenden Zellen und
aktivierten T-Zellen und vermittelt so die Herunterregulierung
von Immunreaktionen, einschließlich der Inhibition der
T-Zellaktivierung und Zytokinproduktion. PD-L1-Expression wird
in Immunzellen und TC beobachtet. Eine aberrante Expression
von PD-L1 auf TC behindert die Antitumor-Immunität, was zu
einer Immunevasion führt. Es wurde festgestellt, dass neue
Immuntherapien, die den PD-L1/PD-1-Signalweg unterbrechen,
die Überlebensraten bei Patienten mit diagnostiziertem NSCLC
verbessern. Die Prognose bei der Behandlung hängt jedoch
vom Grad der PD-L1-Expression ab und erfordert daher die
Quantifizierung von PD-L1 durch immunhistochemische Färbung.
Die Immunhistochemie eignet sich für den Nachweis spezifischer
Antigene in Gewebeproben und ist ein hilfreiches Instrument für
den Pathologen zur Diagnose von Karzinomen und zur Erstellung
einer Prognose. Der VENTANA PD-L1 (SP 263) Assay nutzt einen
monoklonalen Kaninchen-Antikörper zum semiquantitativen Nachweis
von PD-L1-Protein in Schnitten von normalem und neoplastischem
FFPE-Gewebe im Labor. Histologische Gewebepräparate haben den
Vorteil einer intakten Gewebemorphologie und sind daher bei der
Auswertung der PD-L1-Positivität der Patientenprobe hilfreich. Alle
histologischen Tests sollten von einem Spezialisten für die Morphologie
und/oder Pathologie von NSCLC ausgewertet werden. Die Ergebnisse
sollten durch morphologische Studien und geeignete Kontrollen
ergänzt und in Verbindung mit weiteren klinischen Daten und
Labordaten verwendet werden.
4 Algorithmus-Handbuch für die uPath PD-L1 (SP263) Bildanalyse bei nicht-kleinzelligem Lungenkarzinom (Non-Small Cell Lung Cancer, NSCLC)

Testprinzip
Die uPath enterprise software mit dem uPath PD-L1 (SP263)
Bildanalyse-Algorithmus für NSCLC ermittelt einen TC-
Positivitäts-Score auf der Grundlage von Bildanalysetechniken.
Der uPath PD-L1 (SP263) Bildanalyse-Algorithmus für NSCLC
verwendet vorab definierte Parameter zur Bewertung von Bildern
von Gewebe, das mit dem VENTANA PD-L1 (SP263) Assay gefärbt
worden ist.
Bildanalyseschritte:
• Identifizierung von Leerräumen („White Space“) und
automatischer Ausschluss aus der Analyse.
• Erkennung von Zellen im Gesamtbild.
• Einstufung von Zellen als TC oder andere Zelltypen.
• Identifizierung von gefärbten und ungefärbten TC.
• Berechnung des TC-Positivitäts-Scores durch Dividieren der
Anzahl der gefärbten TC durch die Gesamtzahl der TC gemäß
dem Methodenblatt für den VENTANA PD-L1 (SP263) Assay.
Erkennung von TC durch den uPath PD-L1 (SP263)
Bildanalyse-Algorithmus für NSCLC und Berechnung des
Scores:
• Der uPath PD-L1 (SP263) Bildanalyse-Algorithmus für NSCLC
erkennt TC an der Farbe, der Intensität, der Größe und auf der
Grundlage morphologischer Aspekte.
• Die Einstufung der identifizierten TC als gefärbt erfolgt
auf Grundlage der festgestellten Membranstrukturen
und der vorab festgelegten Schwellenwerte gemäß dem
Methodenblatt für den VENTANA PD-L1 (SP263) Assay.
• Zur Berechnung des prozentualen Anteils der gefärbten TC
wird die Anzahl der gefärbten TC gemäß dem Methodenblatt
für den VENTANA PD-L1 (SP263) Assay durch die Gesamtzahl
der TC dividiert.
Erkennung von Leerräumen („White Spaces“) durch den
uPath PD-L1 (SP263) Bildanalyse-Algorithmus für NSCLC:
• Der Algorithmus schließt Leerräume („White Spaces“)
automatisch aus. Artefakte wie Schmutz, Kratzer oder Tinte
werden eventuell nicht automatisch ausgeschlossen.
• Der Anwender kann die vom Algorithmus als Leerräume
bestimmten Bereiche mithilfe eines Falschfarben-Overlays
überprüfen (siehe „PD-L1-Bildanalyse: Falschfarben-Overlay“).
Kriterien des uPath PD-L1 (SP263) Bildanalyse-Algorithmus
für NSCLC zur Ermittlung des TC-Positivitäts-Scores:
• Der uPath PD-L1 (SP263) Bildanalyse-Algorithmus für NSCLC
gibt einen Score mit einer Dezimalstelle an, z.B. „4,8%“.
• Es wird nicht empfohlen, den Score auf- oder abzurunden.
So sollte „4,8%“ beispielsweise nicht auf 5% aufgerundet
werden.
Algorithmus-Handbuch für die uPath PD-L1 (SP263) Bildanalyse bei nicht-kleinzelligem Lungenkarzinom (Non-Small Cell Lung Cancer, NSCLC) 5

Der uPath PD-L1 (SP263) Bildanalyse-Algorithmus für NSCLC
ist zur Verwendung mit dem VENTANA PD-L1 (SP263) Assay
bestimmt. Die Testergebnisse sind immer nur so gut wie die
Qualität und Genauigkeit des dargestellten IHC-Objektträgers
und des analysierten Bildes.
Der Färbelauf mit dem VENTANA PD-L1 (SP263) Assay muss
durch manuelle mikroskopische Untersuchung der PD-L1-
Kontrollobjektträger validiert werden, um zu bestätigen, dass
die erhaltenen Ergebnisse den Erwartungen entsprechen, bevor
Bilder von Objektträgern in die uPath enterprise software geladen
und analysiert werden.
Es sind die Herstellerempfehlungen zur Verwendung
des VENTANA PD-L1 (SP263) Assays einzuhalten,
darunter zur Verwendung aller positiven und negativen
Qualitätskontrollmaterialien bei jedem Färbelauf. Sind die
Ergebnisse der Kontrollobjektträger bei der manuellen
mikroskopischen Untersuchung nicht akzeptabel, dann muss das
Gewebe erneut gefärbt und die Ergebnisse müssen auf Akzeptanz
geprüft werden.
Der Pathologe muss die Empfehlungen zur Auswertung des
VENTANA PD-L1 (SP263) Assay befolgen.
Hinweise dazu finden Sie im Methodenblatt für den VENTANA
PD-L1 (SP263) Assay (Bestell-Nr. 1014258DE) und in der
Interpretationsanleitung (Bestell-Nr. 1015317EN) (verfügbar unter
www.ventana.com).
Der uPath PD-L1 (SP263) Bildanalyse-Algorithmus für NSCLC
ist für die Verwendung durch qualifizierte Pathologen im
Zusammenhang mit den histologischen Untersuchungen,
relevanten klinischen Daten und geeigneten Kontrollen bestimmt.
Er ist kein eigenständiges Tool und erfordert den Eingriff durch
geschulte Personen während des Analyseprozesses.
Der uPath PD-L1 (SP263) Bildanalyse-Algorithmus für NSCLC
kann falsche Ergebnisse liefern, wenn die erfassten Gewebe
anomal gefärbt sind (z.B. die Zellkerne, das Zytoplasma usw.).
Längliche Tumorzellkerne werden vom uPath PD-L1 (SP263)
Bildanalyse-Algorithmus für NSCLC, unabhängig von der Zellform
insgesamt, von der Berechnung ausgeschlossen. Aus diesem
Grund müssen Tumore mit vielen Zellen mit länglichen Zellkernen
unter Umständen manuell ausgewertet werden.
Einschränkungen
Bei der Entwicklung, Programmierung und Validierung des uPath
PD-L1 (SP263) Bildanalyse-Algorithmus für NSCLC wurden
Proben von NSCLC-Gewebe verwendet.
Die Verwendung des uPath PD-L1 (SP263) Bildanalyse-
Algorithmus für NSCLC auf einem Personal Computer (PC) von zu
Hause aus wurde nicht geprüft, und die Sicherheit und Effektivität
einer solchen Verwendung wurden nicht validiert.
Der uPath PD-L1 SP263 Bildanalyse-Algorithmus ist als
Unterstützung bei der Identifizierung von Patienten mit NSCLC zur
Behandlung mit Therapien bestimmt, bei denen der zugelassenen
Fachinformation des therapeutischen Produkts entsprechend für
TC ein PD-L1-Positivitätsgrenzwert von 50% besteht.
Es besteht die Möglichkeit, dass der uPath PD-L1 (SP263)
Bildanalyse-Algorithmus für NSCLC Zellen aus folgenden Gründen
falsch identifiziert: aufgrund einer schwachen Färbung des
Zytoplasmas und/oder der Membran, aufgrund einer starken Färbung
von Immunzellen, welche die Tumorzellfärbung überlagert, wenn
in dem Gewebe gleichzeitig eine erhebliche Entzündung vorliegt,
aufgrund von TC mit mäßig starker, diffuser Zytoplasmapositivität
(„zytoplasmatischer Blush“) und bei unspezifischer Färbung. Dies
kann dazu führen, dass fälschlicherweise TC nicht als TC oder andere
Zellen als positive TC erkannt werden.
Es ist zwar erforderlich, dass Makrophagen aus dem
Analysebereich ausgeschlossen werden, aber dies ist nicht immer
in vollem Umfang möglich. Infolgedessen kann der vom uPath
PD-L1 (SP263) Bildanalyse-Algorithmus für NSCLC ermittelte
Score durch in der zu analysierenden ROI noch vorhandene
Makrophagen beeinflusst werden. Dies ist kritisch, wenn ein
Patientenwert nahe am Grenzwert von 50% liegt.
Die zytoplasmatische Färbung ist im Allgemeinen diffus, weist
aber mitunter eine feine granuläre Qualität auf. In seltenen
Fällen tritt eine perinukleäre punktförmige Körperfärbung mit
unterschiedlicher Intensität auf. Der gesamte prozentuale Anteil
an Membransignalintensitäten im Tumor wird visuell geschätzt
und für die Ermittlung des PD-L1-Expressionsniveaus verwendet.
Die zytoplasmatische Färbung der Tumorzellen wird für die
Bestimmung der PD-L1-Expression ignoriert. Es wird ein mit
dem Isotyp übereinstimmender, negativer Kontrollantikörper
verwendet, um den vorhandenen Hintergrund in Testproben zu
bewerten und eine Baseline für die Färbeintensität festzulegen.
6 Algorithmus-Handbuch für die uPath PD-L1 (SP263) Bildanalyse bei nicht-kleinzelligem Lungenkarzinom (Non-Small Cell Lung Cancer, NSCLC)

Datensicherheit
Schadsoftware und unbefugte Zugriffe können zu Datenverlusten
und Nichtverfügbarkeit des Geräts führen.
Um eine Infektion durch Schadsoftware, unbefugte Zugriffe und
Gerätemissbrauch zu vermeiden, sind folgende Empfehlungen
wesentlich:
• Installieren und führen Sie keine andere Software auf dem
Gerät aus.
• Stellen Sie sicher, dass andere Rechner und Dienste
im Netzwerk ordnungsgemäß gesichert und gegen
Schadsoftware und unbefugte Zugriffe geschützt sind. Dies
betrifft zum Beispiel das Labor-Informationssystem (LIS) und
Archivierungs- und Backup-Laufwerke oder -Dienste.
• Die Kunden sind für die Sicherheit ihres lokalen
Netzwerks verantwortlich, vor allem beim Schutz gegen
Schadsoftware und Angriffe. Dieser Schutz kann auch
Maßnahmen wie eine Firewall beinhalten, um das Gerät von
unkontrollierten Netzwerken zu trennen, und Maßnahmen,
die sicherstellen, dass das angeschlossene Netzwerk frei von
Schadsoftwarecode ist.
• Beschränken Sie den physischen Zugriff auf das Gerät und
alle angeschlossenen IT-Infrastrukturen (Rechner, Kabel,
Netzwerkgeräte usw.).
• Stellen Sie sicher, dass die Geräte-Backup- und Archivdateien
vor unbefugten Zugriffen und Zerstörung geschützt sind.
Diese Liste umfasst auch den entfernten Speicherort,
Disaster-Recovery-Speicherorte und die sichere Übertragung
von Backup-Dateien.
• Verwenden Sie nach Möglichkeit eine Firewall, um den
Netzwerkverkehr zu beschränken.
• USB-Flash-Speichermedien eignen sich für verschiedene
Daten-Backups und -Wiederherstellungen. Ein
unsachgemäßer Umgang mit USB-Flash-Speichermedien
kann zu Datenverlusten oder Gerätestörungen führen.
• Verwenden Sie ausschließlich USB-Flash-Speichermedien,
die vom Roche Servicetechniker getestet und installiert
wurden.
• Zu jedem Zeitpunkt kann nur ein USB-Speichermedium
gleichzeitig verwendet werden. Bevor Sie ein USB-Flash-
Speichermedium einstecken, sollten Sie daher prüfen, ob
bereits andere USB-Speichermedien angeschlossen sind.
• Vor dem Herausziehen eines USB-Flash-Speichermediums
wählen Sie in Windows die Schaltfläche „Auswerfen“.
• Die Betriebssystem(OS)-Standardkonfiguration des Servers
bei der Lieferung darf nicht geändert werden, da dies
Auswirkungen auf die aus Sicherheitsgründen gehärtete OS-
Konfigurationen hätte.
• Um eine Virusinfektion der uPath enterprise software zu
vermeiden, verwenden Sie das USB-Flash-Speichermedium
nur bei diesem Gerät. Speichern Sie keine anderen Daten auf
diesem USB-Flash-Speichermedium.
Algorithmus-Handbuch für die uPath PD-L1 (SP263) Bildanalyse bei nicht-kleinzelligem Lungenkarzinom (Non-Small Cell Lung Cancer, NSCLC) 7

Arbeitsablauf mit dem uPath PD-L1 Bildanalyse-Algorithmus für NSCLC
Benötigte Materialien
• uPath enterprise software
• uPath PD-L1 (SP263) Bildanalyse-Algorithmus für NSCLC
• Objektträger mit NSCLC-Gewebe, gefärbt mit dem VENTANA PD-L1 (SP263) Assay (unter Verwendung des OptiView DAB IHC
Detection Kit) auf dem BenchMark ULTRA-Gerät
• VENTANA DP 200 Objektträger-Scanner
Arbeitsablauf
1. Eine NSCLC-Gewebeprobe auf einem Glas-Objektträger wird mit dem VENTANA PD-L1 (SP263) Assay in einem BenchMark
ULTRA-Gerät gefärbt.
2. Die Bilderfassung (Gesamtobjektträger-Scan) erfolgt mit einem VENTANA DP 200 Objektträger-Scanner bei 20facher
Vergrößerung in einer z-Ebene.
3. Die erfassten digitalen Bilder werden von dem Computer, der mit dem VENTANA DP 200 Objektträger-Scanner verbunden ist, zum
Bildverwaltungssystem (IMS) auf einen zentralen Server übertragen.
4. Nach dem Transfer zum Server legt die uPath enterprise software einen Fall an. Die Fallerstellung kann automatisch und durch
Kommunikation mit dem Laborinformationssystem (LIS) erfolgen: Dabei werden die ID-Daten (z.B. Gewebetyp und primärer
Antikörper) auf dem Barcode-Etikett des Objektträgers verwendet. Alternativ werden die Daten manuell in die uPath enterprise
software eingegeben (siehe das Benutzerhandbuch für die uPath enterprise software [Bestell.-Nr. 1018943DE]).
5. Ist der uPath PD-L1 (SP263) Bildanalyse-Algorithmus für NSCLC auf einem anderen Server als die uPath enterprise software und
das IMS installiert, und wird ein 20fach vergrößertes Bild mit dem passenden Färbe- und Gewebetyp aufgenommen, veranlasst die
uPath enterprise software automatisch eine Gesamtobjektträger-Analyse (WSA, Whole Slide Analysis).
6. Im Rahmen der WSA wird das gescannte Gesamtbild automatisch analysiert.
7. Nach Abschluss der WSA erhält der Pathologe in der uPath enterprise software folgende Meldung: „analysis is complete“ (Analyse
abgeschlossen). Nun kann der Pathologe bestimmte ROIs auswählen und mit Scores bewerten. Es können eine einzelne ROI
beliebiger Größe oder mehrere ROIs ausgewählt werden. Bei Auswahl mehrerer ROIs werden sowohl ein zusammenfassender
Score als auch individuelle Scores für jede ROI ausgegeben.
Färbung
• Die Vorbereitung und Färbung der Gewebe sollten gemäß den Empfehlungen im Methodenblatt für den VENTANA PD-L1 (SP263)
Assay erfolgen.
• Es sind alle erforderlichen Kontrollen zu prüfen. Entspricht die Färbung nicht den Empfehlungen im Methodenblatt für den
VENTANA PD-L1 (SP263) Assay, muss sie wiederholt werden.
• Vor der Analyse mit dem uPath PD-L1 (SP263) Bildanalyse-Algorithmus für NSCLC muss das Gewebe mit dem VENTANA PD-
L1 (SP263) Assay unter Verwendung aller im Methodenblatt für den VENTANA PD-L1 (SP263) Assay aufgeführten zusätzlichen
Materialien bzw. Zubehörartikel gefärbt werden. Der VENTANA PD-L1 (SP263) Assay erlaubt den Nachweis von PD-L1-Protein in
FFPE-NSCLC-Gewebe nach Färbung mit dem OptiView DAB IHC Detection Kit auf einem BenchMark ULTRA-Gerät. Obwohl mit
dem VENTANA PD-L1 (SP263) Assay PD-L1-Protein in FFPE-NSCLC-Gewebe nach Färbung auf einem BenchMark ULTRA-Gerät
nachgewiesen wird, wurde der uPath PD-L1 (SP263) Bildanalyse-Algorithmus für NSCLC nach Färbung mit dem OptiView DAB IHC
Detection Kit auf einem BenchMark ULTRA-Gerät validiert.
Bilderfassung
Für das Scannen der Objektträger ist ein VENTANA DP 200 Objektträger-Scanner erforderlich. Die Bilder werden bei 20facher
Vergrößerung aufgenommen. Wenn größere Abschnitte des Bildes nicht scharf sind, wird empfohlen, die Objektträger ein zweites Mal
zu scannen. Weiterführende Hinweise zum Scannen finden Sie im Benutzerhandbuch für den VENTANA DP 200 Objektträger-Scanner
(Bestell-Nr.1017149DE).
8 Algorithmus-Handbuch für die uPath PD-L1 (SP263) Bildanalyse bei nicht-kleinzelligem Lungenkarzinom (Non-Small Cell Lung Cancer, NSCLC)

Allgemeine Navigation: uPath enterprise software
Die uPath enterprise software erlaubt eine Anpassung an die Anforderungen einzelner Personen und Standorte, was insbesondere
die Berichtkonfiguration und die Benutzeroberfläche betrifft. Dieses Algorithmus-Handbuch konzentriert sich ausschließlich auf die
Werkzeuge, die für den uPath PD-L1 (SP263) Bildanalyse-Algorithmus für NSCLC benötigt werden. Weiterführende Hinweise zur uPath
enterprise software finden Sie im Benutzerhandbuch für die uPath enterprise software.
Arbeitsablauf für Pathologen
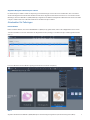
Einen Fall öffnen
Bilder von NSCLC-Gewebe, das mit dem VENTANA PD-L1 (SP263) Assay gefärbt wurde, können durch Doppelklicken auf einen Fall
oder durch Auswählen eines Falls und Drücken der Registerkarte Viewer (Anzeige) in der uPath enterprise software geöffnet werden
(Abb. 1).
Es wird ein Bildschirm mit allen Bildern angezeigt, die mit dem Fall assoziiert sind (Abb.2).
Abb.2
Abb.1
Algorithmus-Handbuch für die uPath PD-L1 (SP263) Bildanalyse bei nicht-kleinzelligem Lungenkarzinom (Non-Small Cell Lung Cancer, NSCLC) 9

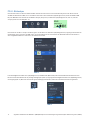
Nach der Färbung mit dem VENTANA PD-L1 (SP263) Assay wird der Objektträger mit dem VENTANA DP 200Objektträger-Scanner bei
20facher Vergrößerung eingescannt. Das Bild wird von der uPath enterprise software importiert und einem Fall zugewiesen. Der uPath PD-L1
(SP263) Bildanalyse-Algorithmus für NSCLC löst automatisch eine WSA aus. Wie viel Zeit die WSA-Vorberechnungen in Anspruch nehmen,
hängt von den Serverspezifikationen, der Bildgröße und der Anzahl der Bilder in der Warteschlange ab. Die Meldung „waiting to start auto-
analysis” (Wartet auf Start der automatischen Analyse) bedeutet, dass sich die Bilder in der Warteschlange befinden, aber noch nicht analysiert
wurden; „analyzing” (Analyse läuft) bedeutet, dass gerade eine WSA durchgeführt wird (Abb.3 und 4). Nach Abschluss der Bild-WSA durch die
uPath enterprise software wird im Viewer (Anzeige) der uPath enterprise software die Meldung „analysis successful“ (Analyse erfolgreich) unter
dem Objektträgerbild angezeigt (Abb. 5). Die Punktbewertung kann erst nach Abschluss einer erfolgreichen WSA durchgeführt werden.
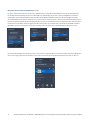
Gesamttumor-ROI(s) kennzeichnen: Tumorbereich auswählen
Wählen Sie im ROI-Dropdown-Menü (Abb.6) die Schaltfläche Freehand (Freihandwerkzeug) aus und wählen Sie mit diesem Werkzeug
auf dem IHC-Objektträgerbild den Tumorbereich/die Tumorbereiche aus, der/die analysiert werden soll/en. In Abb.7 ist ein Bild
dargestellt, bei dem eine einzelne ROI gekennzeichnet wurde. Es können zusätzliche ROIs gekennzeichnet werden. Jeder ausgewählte
Bereich führt dazu, dass im Slide Panel (Objektträgerbereich) eine ROI angezeigt wird (Abb.8).
Abb.6 Abb.7
Abb.8
Abb.5
Abb.4
Abb.3
10 Algorithmus-Handbuch für die uPath PD-L1 (SP263) Bildanalyse bei nicht-kleinzelligem Lungenkarzinom (Non-Small Cell Lung Cancer, NSCLC)

Gesamttumor-ROI(s) kennzeichnen: Ausschlussbereich
Beim Kennzeichnen von ROIs kann es notwendig sein, bestimmte Bereiche auszuschließen. Bereiche, die besser vermieden bzw.
ausgelassen werden, und Beispiele für solche Bereiche werden im Abschnitt „Färbungsmerkmale“ beschrieben. Im Dropdown-Menü
Exclusion (Ausschluss) (Abb. 9) wählen Sie Freehand (Freihandwerkzeug), um bestimmte Bereiche auszuschließen (Abb.10). Sind
große Bildbereiche verschwommen bzw. nicht scharf, scannen Sie den Objektträger ein zweites Mal.
Ausgeschlossene Bereiche werden von dem Algorithmus nicht berücksichtigt. Gefärbte und ungefärbte TC in diesem Bereich werden
von der Gesamtanalyse ausgeschlossen. Wird ein Ausschlussbereich bei einer ROI definiert, die bereits analysiert worden ist, muss die
ROI erneut analysiert werden. Die Überlappung und der Score werden entsprechend aktualisiert.
Die Kennzeichnung einer großen Anzahl von Ausschlussregionen, insbesondere komplizierter Ausschlüsse mit Freehand
(Freihandwerkzeug), kann zeitaufwändig sein und die Effizienz des Arbeitsablaufs beeinträchtigen, wirkt sich aber nur geringfügig auf
das Endergebnis aus. Wenn ein Fall eine hohe Anzahl von Ausschlüssen erfordert, wird folgende Vorgehensweise empfohlen:
• Kennzeichnen Sie mehrere ROIs und schließen Sie unter minimalem Einsatz des Werkzeugs Exclusion (Ausschluss) Teile des
Gewebes aus, die Sie als nicht auswertbar erachten.
• Begrenzen Sie die Zahl der Ausschlüsse und überschreiben Sie den Score mit einem anderen Score.
Gesamttumor-ROI(s) kennzeichnen: Löschen
Ist die ausgewählte Gesamttumor-ROI nicht optimal, kann sie gelöscht werden. Klicken Sie auf die Mitte der Gesamttumor-ROI auf dem
Bild, um sie auszuwählen. Klicken Sie dann im Slide Panel (Objektträgerbereich) auf die Schaltfläche Delete (Löschen) (Abb. 11) oder
auf das Abfalleimersymbol im Objektträgerbild neben der ROI (Abb. 12). Es wird ein Bestätigungsfenster angezeigt. Wählen Sie Confirm
(Bestätigen), um die Löschung der ausgewählten ROI zu bestätigen. Wählen Sie Cancel (Abbrechen), um die ROI beizubehalten.
Abb.10Abb.9
Abb.11 Abb.12
Algorithmus-Handbuch für die uPath PD-L1 (SP263) Bildanalyse bei nicht-kleinzelligem Lungenkarzinom (Non-Small Cell Lung Cancer, NSCLC) 11

Nach dem Kennzeichnen aller Gesamttumor-ROIs und/oder Ausschlussbereiche kann das Bild analysiert werden. Klicken Sie auf
die Mitte der Gesamttumor-ROI, um sie auszuwählen oder klicken Sie im Slide Panel (Objektträgerbereich) auf die Schaltfläche ROI.
Bei jeder ROI wählen Sie entweder die Schaltfläche Analyze (Analysieren) im Slide Panel (Objektträgerbereich, Abb.13) oder das
Analysesymbol neben der ROI (Abb.14).
Nach Abschluss der PD-L1-Analyse wird das Ergebnis an zwei Stellen im Slide Panel (Objektträgerbereich) angezeigt: unter Slide Score
(Objektträger-Score) und neben den ROIs (Abb.15). Der Objektträger-Score ist die Summe des Positivitätsstatus der Tumorzellen in
allen ausgewählten ROIs. Dies ist der Score, der im Bericht erscheint.
PD-L1-Bildanalyse
Abb.15
In den Ausklappbereichen Slide Score (Objektträger-Score) und ROI Details (ROI-Details) finden Sie weiterführende Informationen. Diese
Bereiche werden durch Klicken auf das jeweilige Ausklappsymbol (Abb.16) angezeigt. Der Ausklappbereich Slide Score (Objektträger-Score)
wird angezeigt (Abb.17). Wenn Sie erneut auf das gleiche Ausklappsymbol klicken, werden diese Informationen wieder ausgeblendet.
Abb.17
Abb.16
Abb.13 Abb.14
12 Algorithmus-Handbuch für die uPath PD-L1 (SP263) Bildanalyse bei nicht-kleinzelligem Lungenkarzinom (Non-Small Cell Lung Cancer, NSCLC)

PD-L1-Bildanalyse: Falschfarben-Overlay
Nach Drücken der Schaltfläche ROI(s) Analysis (ROI-Analyse) und dem Abschluss der Gewebeanalyse wird auf der ROI ein Farb-
Overlay angezeigt. In der Abbildung unten (Abb.18) stellen die roten Kreise Zellen dar, bei denen positive PD-L1-Färbung festgestellt
wurde. Die blauen Kreise stellen Zellen dar, bei denen festgestellt wurde, dass sie für PD-L1 negativ sind. Wird das Bild bewegt (durch
Drücken der linken Maustaste und Ziehen des Bilds), verschwindet das Overlay (Abb.19). Wenn die Maustaste losgelassen wird, wird
das Overlay wieder eingeblendet (Abb.18).
Abb.18
Abb.19
Algorithmus-Handbuch für die uPath PD-L1 (SP263) Bildanalyse bei nicht-kleinzelligem Lungenkarzinom (Non-Small Cell Lung Cancer, NSCLC) 13

Manuelles Überschreiben des Objektträger-Scores
Die Scores können manuell überschrieben werden. Dazu klicken Sie im Slide Panel (Objektträgerbereich) auf das Ausklappsymbol
für den Slide Score (Objektträger-Score) neben dem Slide Score (Objektträger-Score) (Abb. 16). Der Ausklappbereich Slide Score
(Objektträger-Score) wird angezeigt (Abb.20). Nach Wahl der Schaltfläche Edit (Bearbeiten) (Abb. 20) im Ausklappbereich Slide
Score (Objektträger-Score) kann der Anwender einen manuellen Score eingeben (Abb.21). Im Feld Comments (Kommentare) können
Hinweise zu dem Fall und/oder der Entscheidung zum Überschreiben des automatischen Scores eingegeben werden. Im Fall von PD-L1
können Scores von 0–100% manuell eingegeben werden. Nach Eingabe des manuellen Scores wählen Sie die Schaltfläche Confirm
(Bestätigen), um diese manuelle Überschreibung zu bestätigen (Abb.22). Klicken Sie in der Bestätigungsmeldung auf „Yes” (Ja).
Nun wird im Objektträgerbereich der überschriebene, manuelle Score angezeigt. Neben den betroffenen ROIs wird nun kein Bildanalyse-
Score mehr angezeigt (Abb.23). Um das Bild erneut zu analysieren, drücken Sie auf das Balkendiagrammsymbol (Abb.13, Abb.14).
Abb.21 Abb.22
Abb.20
Abb.23
14 Algorithmus-Handbuch für die uPath PD-L1 (SP263) Bildanalyse bei nicht-kleinzelligem Lungenkarzinom (Non-Small Cell Lung Cancer, NSCLC)

Beachten Sie das Methodenblatt für den VENTANA PD-L1 (SP263) Assay und die Interpretationsanleitung.
Bewertung der PD-L1-Färbung
Mit dem VENTANA PD-L1 (SP263) Assay markierte neoplastische NSCLC-Zellen werden auf die prozentuale Positivität der TC
anhand der Membranfärbung jeder Intensität untersucht. Die immunhistochemische Färbung bei NSCLC ist membranär und/oder
zytoplasmatisch, und die Expression kann in der gesamten Neoplasie homogen oder heterogen sein. Die zytoplasmatische Färbung
von Tumorzellen spielt für den TC-Positivitäts-Score keine Rolle. Die Membranfärbung kann ein diskontinuierliches, zirkumferentielles
oder basolaterales Muster aufweisen. Es wird ein Antikörper gleichen Isotyps als Negativkontrolle verwendet, um den vorhandenen
Hintergrund in Testproben zu bewerten.
Bewertung der Färbung mit dem PD-L1-Algorithmus
Der Pathologe, der den uPath PD-L1 (SP263) Bildanalyse-Algorithmus für NSCLC verwendet, sollte mit der manuellen Bewertung
des VENTANA PD-L1 (SP263) Assay vertraut sein. Der Pathologe sollte den Gesamttumorbereich mit dem Freihandwerkzeug
einkreisen. Vor der Verwendung des uPath PD-L1 (SP263) Bildanalyse-Algorithmus für NSCLC sollten die zugehörigen H&E- und
Negativkontrollobjektträger analysiert werden. Bei der Auswahl der zu analysierenden Bereiche sind die in den Abschnitten
„Einschränkungen“ und „Zu meidende Bereiche“ beschriebenen Erwägungen zu beachten. Der vom uPath PD-L1 (SP263) Bildanalyse-
Algorithmus für NSCLC vorgeschlagene automatische Score kann vom Pathologen gegebenenfalls manuell überschrieben werden.
Nicht bewertbare Fälle umfassen, sind jedoch nicht beschränkt auf, Fälle mit zu wenigen lebensfähigen Tumorzellen, inakzeptabler
Morphologie und störendem Hintergrund. NSCLC-Fälle mit genügend vitalen TC (laut Feststellung des bewertenden Pathologen) und
ohne störenden Hintergrund auf dem PD-L1-IHC-Objektträger sind für eine Beurteilung geeignet.
Färbungsmerkmale
Algorithmus-Handbuch für die uPath PD-L1 (SP263) Bildanalyse bei nicht-kleinzelligem Lungenkarzinom (Non-Small Cell Lung Cancer, NSCLC) 15

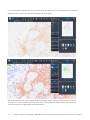
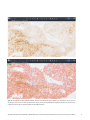
Abb.24: Scan-Bilder von IHC-gefärbtem NSCLC-Gewebe in der uPath enterprise software; vor der Analyse (oben) und nach
der Analyse (unten). Das rote Overlay repräsentiert Zellen, die als positiv gefärbte TC identifiziert wurden. Das blaue Overlay
repräsentiert Zellen, die als negativ gefärbte TC identifiziert wurden.
In den nachfolgenden Abbildungen (Abb.24–25) sind verschiedene Färbemuster und verschiedene Grade der PD-L1-Expression
dargestellt. Die PD-L1-Expression in TC wird als Prozentanteil (0–100) angegeben.
16 Algorithmus-Handbuch für die uPath PD-L1 (SP263) Bildanalyse bei nicht-kleinzelligem Lungenkarzinom (Non-Small Cell Lung Cancer, NSCLC)
Seite laden ...
Seite laden ...
Seite laden ...
Seite laden ...
Seite laden ...
Seite laden ...
Seite laden ...
Seite laden ...
Seite laden ...
Seite laden ...
Seite laden ...
Seite laden ...
Seite laden ...
Seite laden ...
Seite laden ...
Seite laden ...
Seite laden ...
Seite laden ...
-
 1
1
-
 2
2
-
 3
3
-
 4
4
-
 5
5
-
 6
6
-
 7
7
-
 8
8
-
 9
9
-
 10
10
-
 11
11
-
 12
12
-
 13
13
-
 14
14
-
 15
15
-
 16
16
-
 17
17
-
 18
18
-
 19
19
-
 20
20
-
 21
21
-
 22
22
-
 23
23
-
 24
24
-
 25
25
-
 26
26
-
 27
27
-
 28
28
-
 29
29
-
 30
30
-
 31
31
-
 32
32
-
 33
33
-
 34
34
-
 35
35
-
 36
36
-
 37
37
-
 38
38
Roche uPath PD-L1 image analysis for NSCLC IVD Algorithm Benutzerhandbuch
- Typ
- Benutzerhandbuch
Verwandte Papiere
-
Roche VENTANA PD-L1 (SP263) Assay, Universal, CE IVD, Predictive, NSCLC Interpretation Guide
-
Roche uPath IVD Benutzerhandbuch
-
Roche VENTANA anti-ALK D5F3 Primary Antibody Benutzerhandbuch
-
Roche BenchMark ULTRA Benutzerhandbuch
-
Roche uPath IVD Benutzerhandbuch
-
Roche BenchMark XT/LT Interpretation Guide
-
Roche Virtuoso Benutzerhandbuch
-
Roche Virtuoso Benutzerhandbuch
Sonstige Unterlagen
-
IMG Stage Line WSA-50WIFI Bedienungsanleitung
-
 Hologic Genius Digital Diagnostics System Bedienungsanleitung
Hologic Genius Digital Diagnostics System Bedienungsanleitung
-
 Hologic Genius Review Station Bedienungsanleitung
Hologic Genius Review Station Bedienungsanleitung
-
 Hologic ThinPrep Genesis Processor Bedienungsanleitung
Hologic ThinPrep Genesis Processor Bedienungsanleitung
-
 Hologic ThinPrep Integrated Imager Bedienungsanleitung
Hologic ThinPrep Integrated Imager Bedienungsanleitung
-
 Hologic ThinPrep 2000 Processor Bedienungsanleitung
Hologic ThinPrep 2000 Processor Bedienungsanleitung










































